[TOC]
一、前言
最近学习open3d的可视化drr与三维椎体,需要导入三维椎体的.ply格式,源文件是.nii.gz格式,但是mimics不能导入.nii.gz,所以需要先利用将slicer将.nii.gz导出为.dcm格式。然后再用mimics将.dcm导出为.ply格式。本文主要作为个人笔记。
二、步骤
2.1.slicer将.nii.gz格式转为.dcm格式
由于.nii.gz不能直接导入到mimics,需要先利用slicer作为中转站,将.nii.gz导为.dcm文件。本文中我的需求是将四个.nii.gz文件导为一个.dcm文件。
2.1.1导入.nii.gz文件
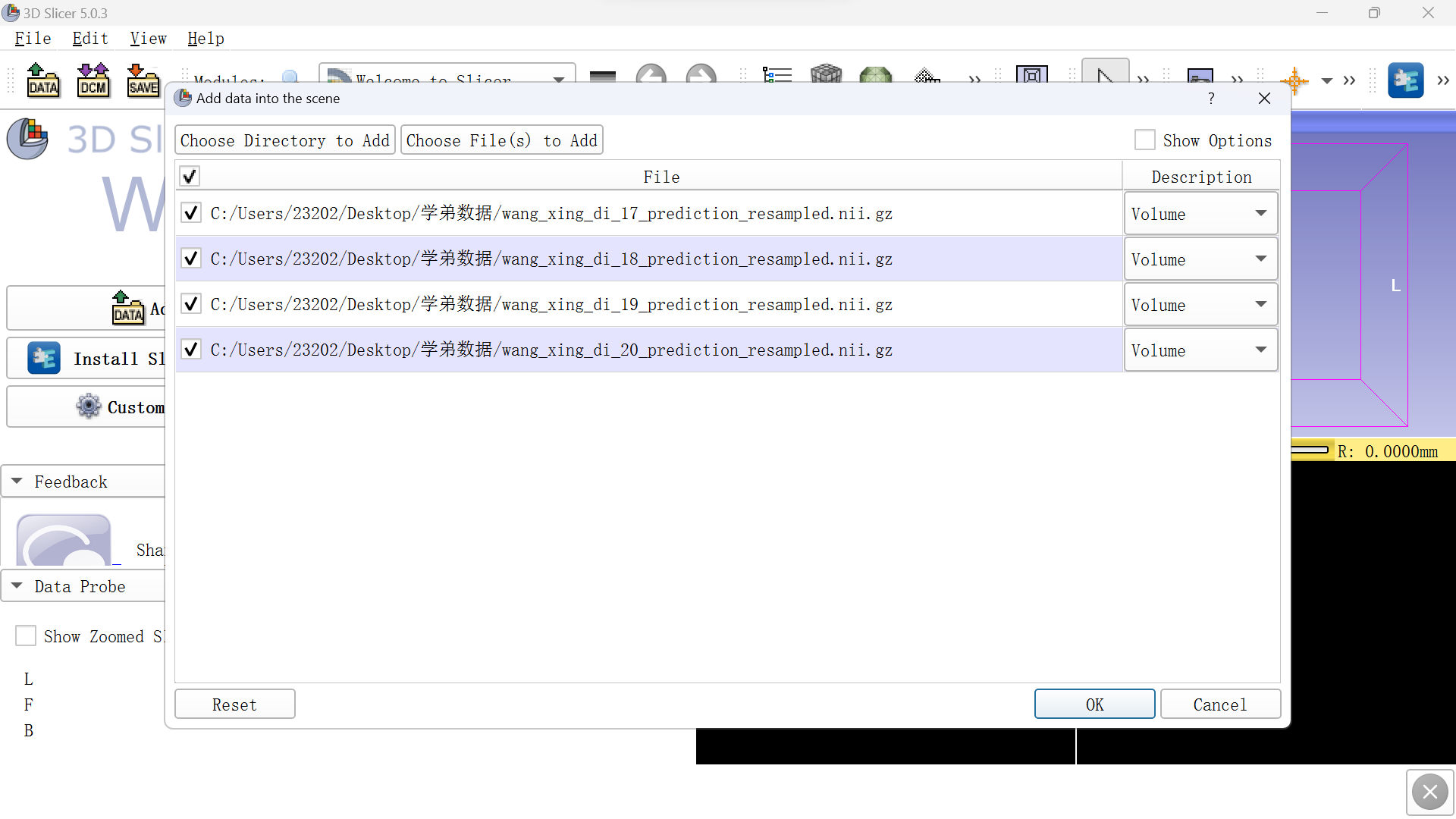
打开slicer,导入四个.nii.gz文件,点击ok。
2.1.2.可视化渲染
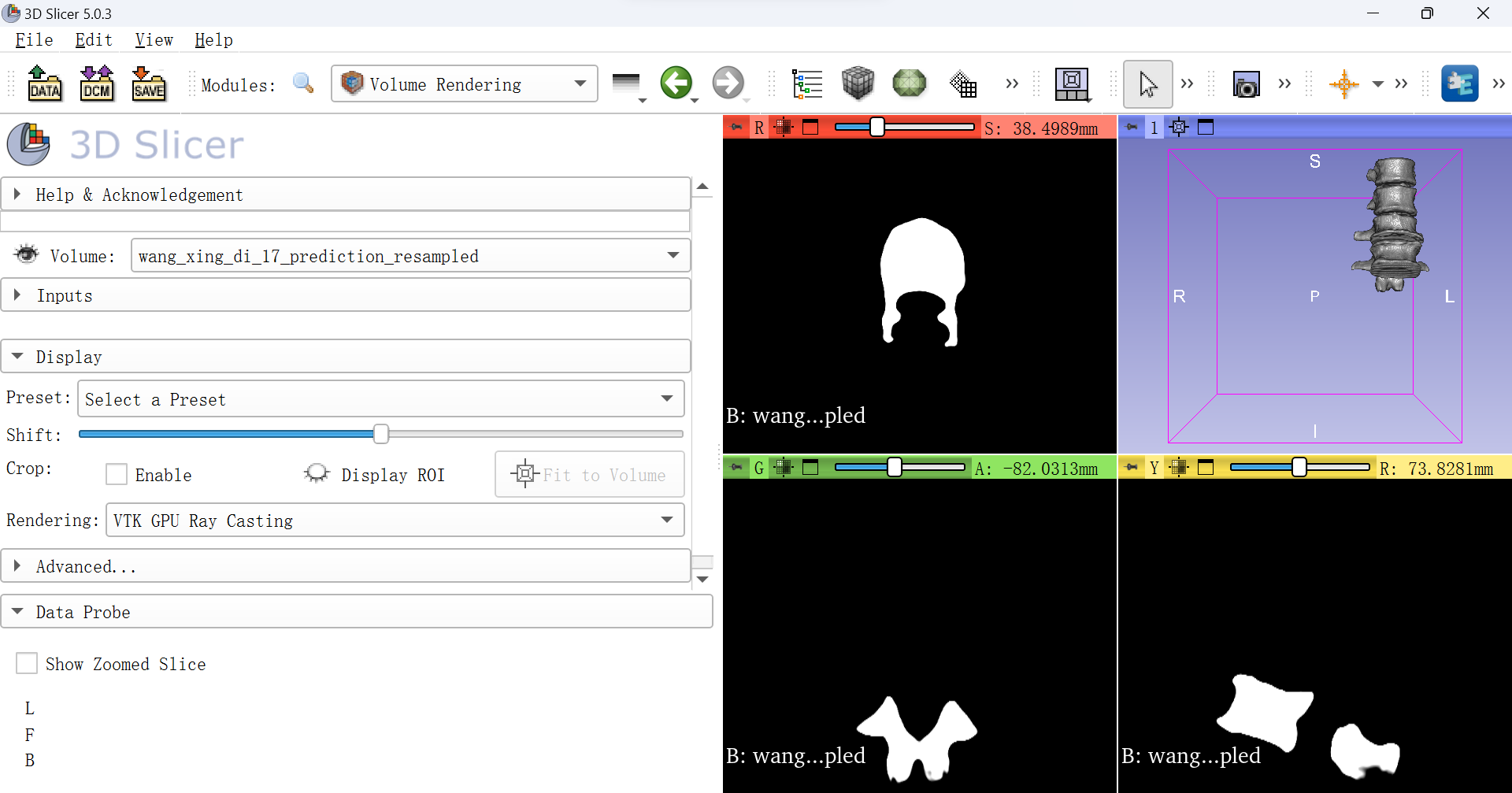
这里我们在Modules那一栏选择Volume Rendering看一下文件,可以看到我这里导入的四个文件(需要在Volume那一栏选择每个文件打开”眼睛“按钮才能看到)
2.1.3.新建一个segmentation
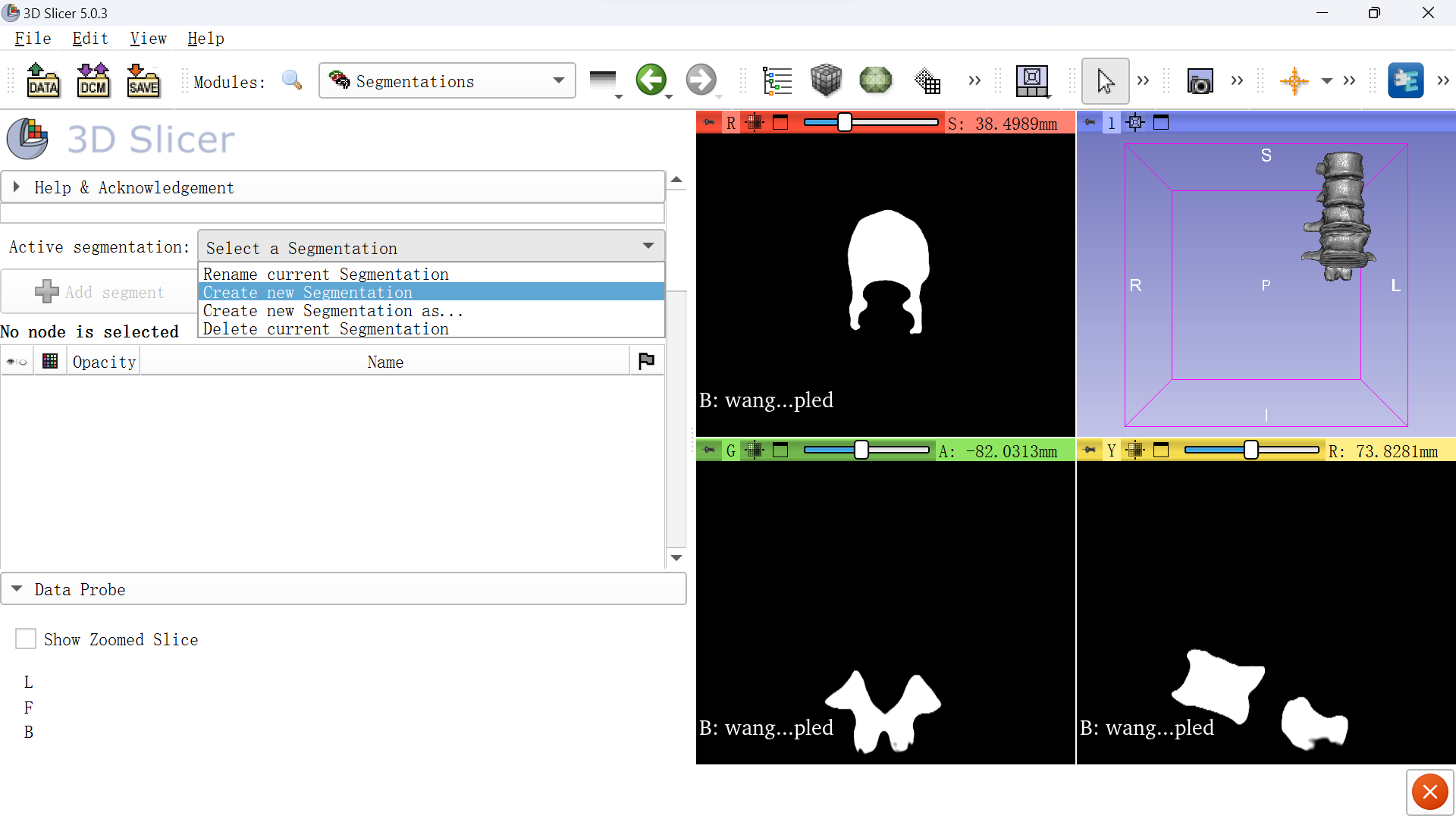
同样在Modules选择Segmentations,然后在Activate segmentation选择create new segmentation,
2.1.4.添加到segmenation
点击完create segmenation后会出现一个笔的符号,点击后就得到如下界面,就可以开始添加了,选择master volume里面的文件,点击Add。
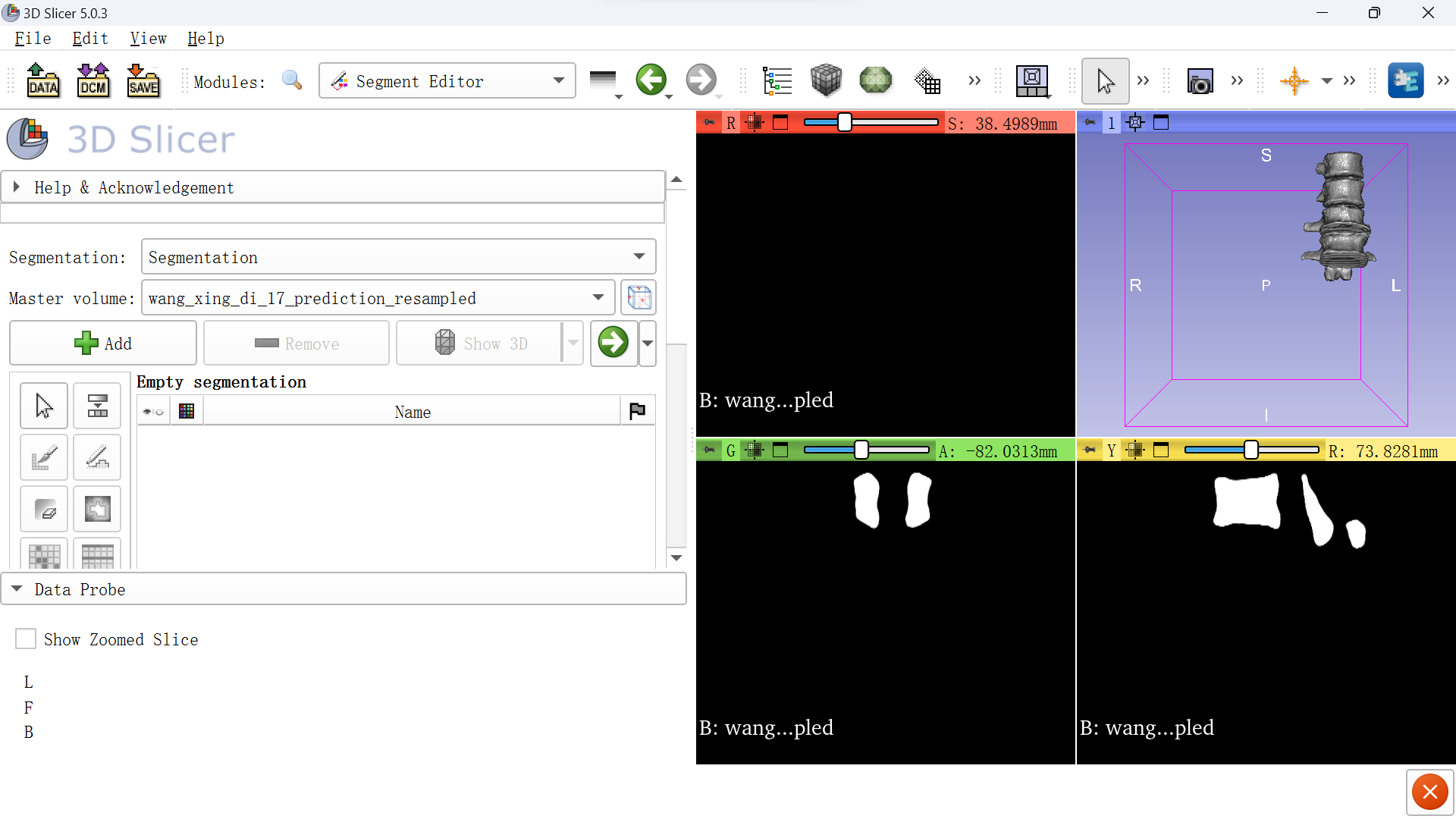
再选择Threshold(就在Add下面,紧挨着)。
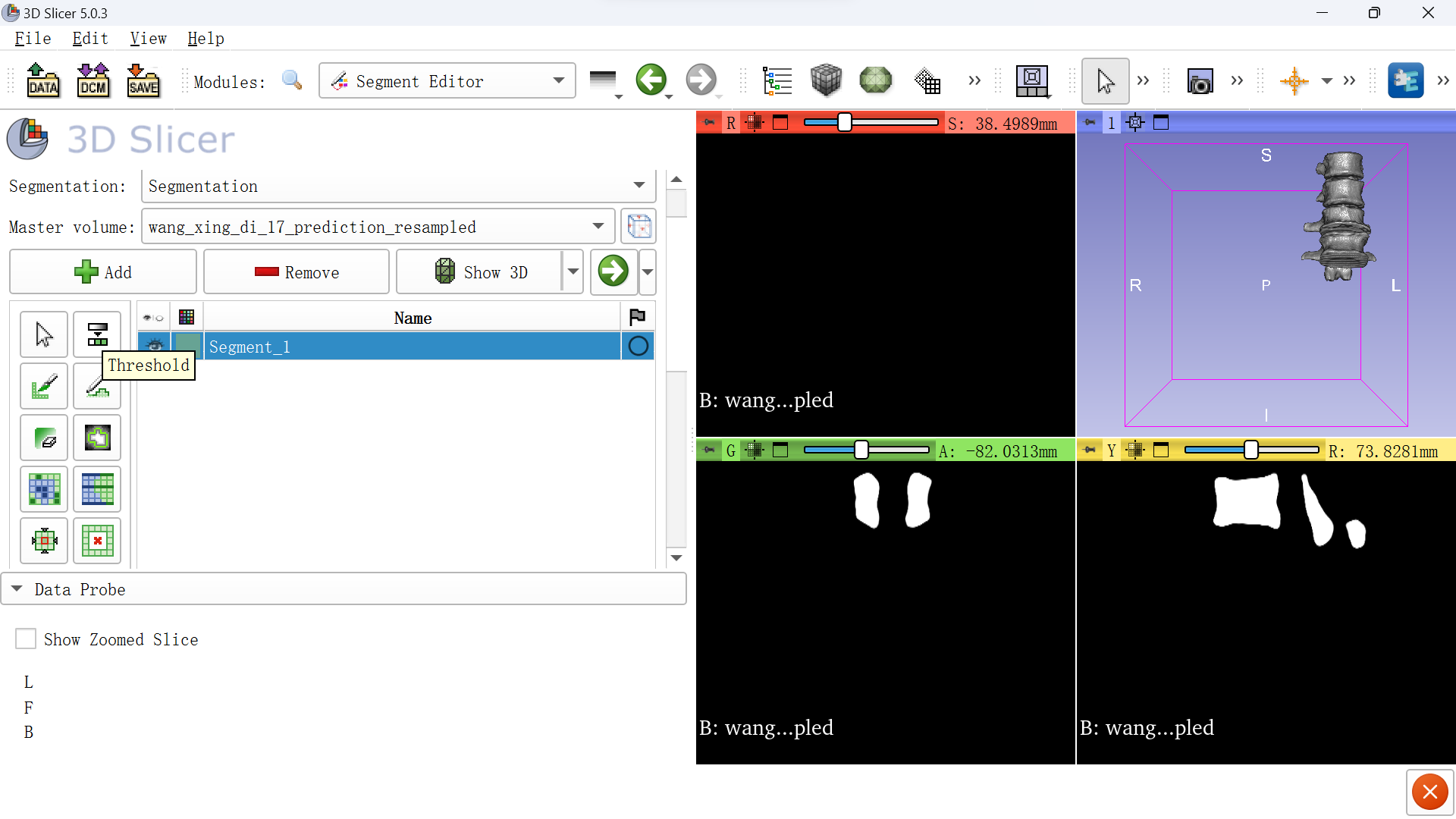
再滑到下面,将进度条拉到最右边,再点击apply。如此一个文件就加进来了,再把剩下的导入就行,操作一样。
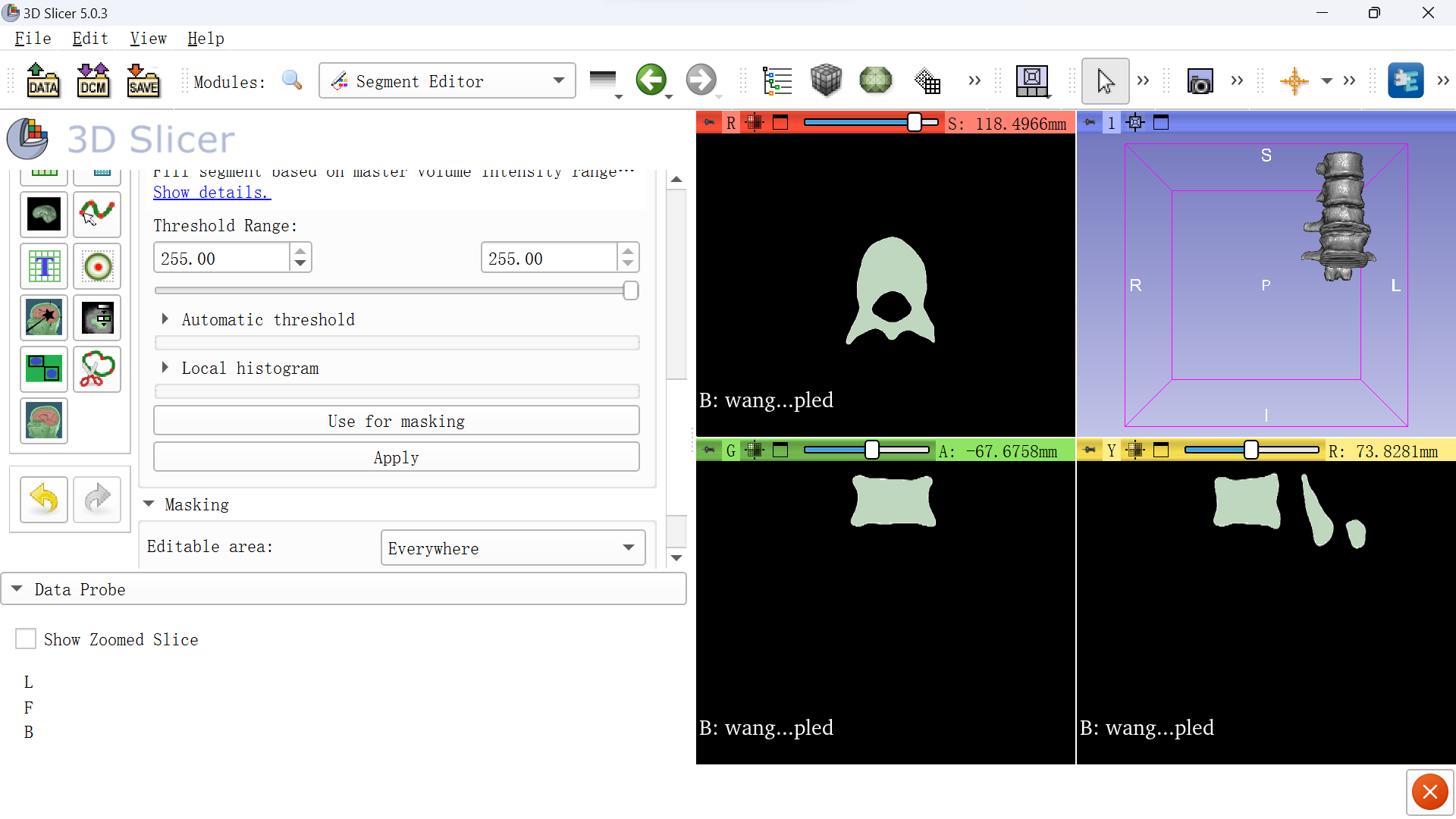
添加完为这样
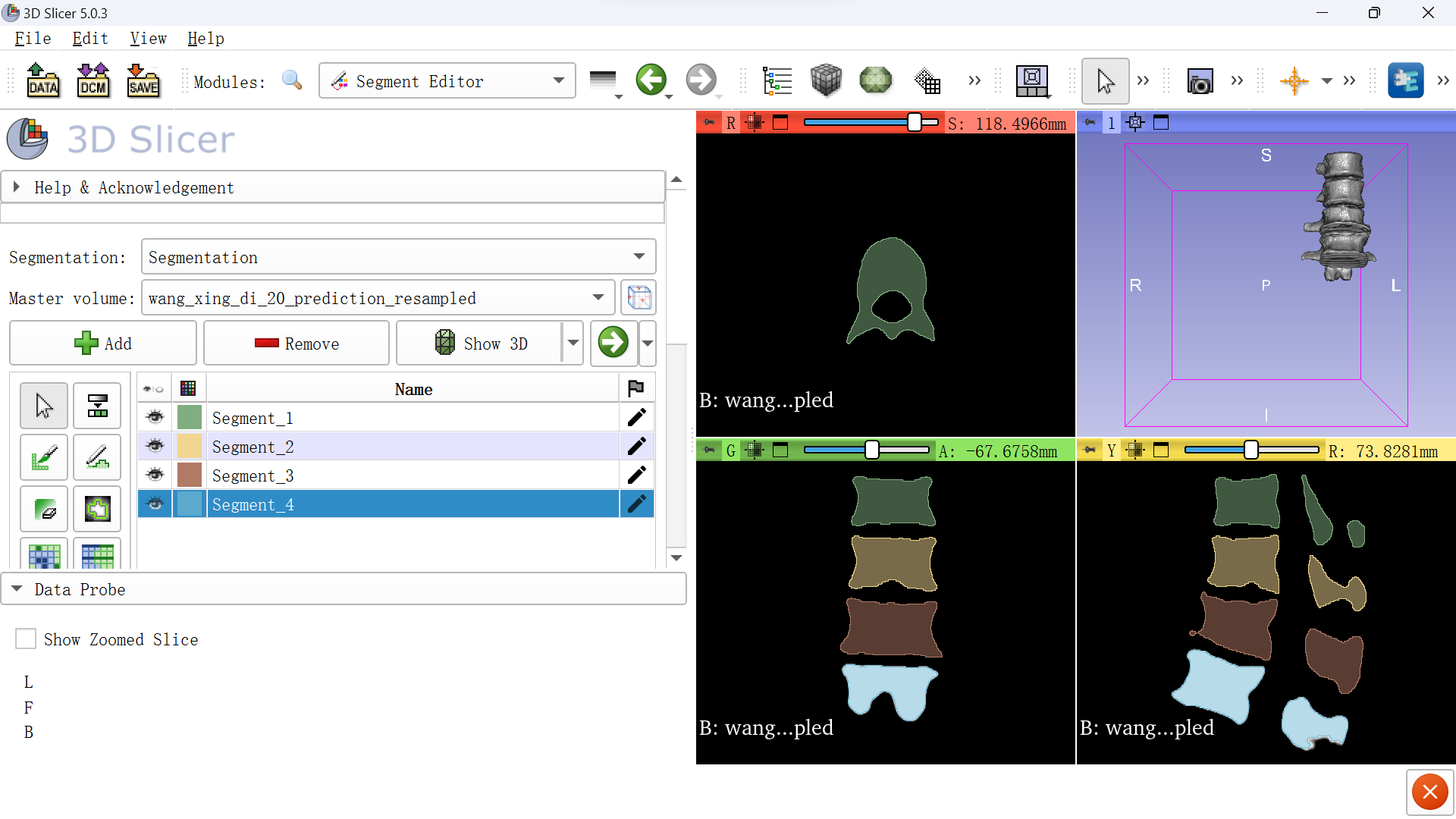
2.1.5.导出为.dcm文件
然后选择Modules为Data。
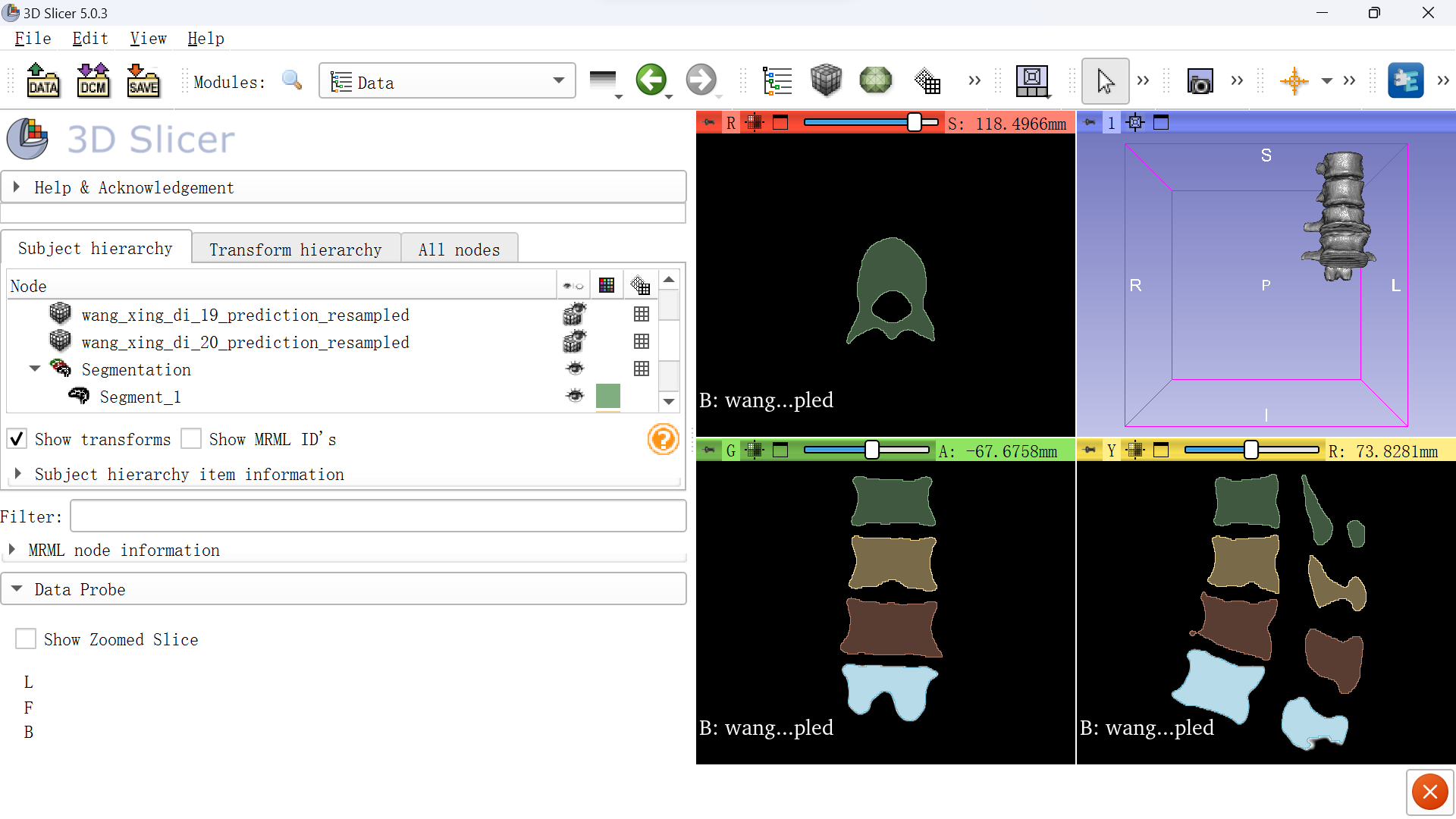
点击Segmenation右键导出为Dicom。
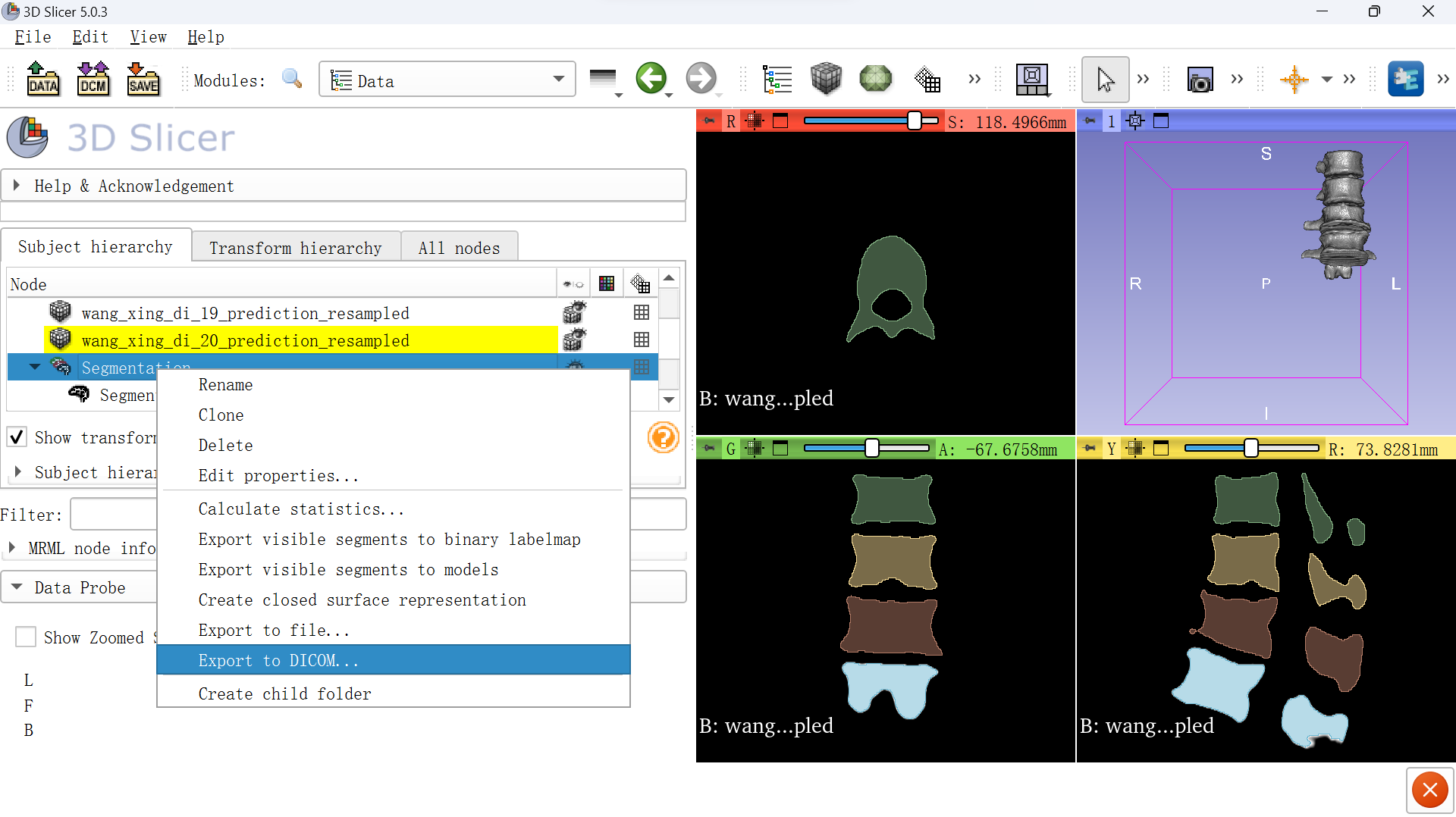
选择文件导出即可。
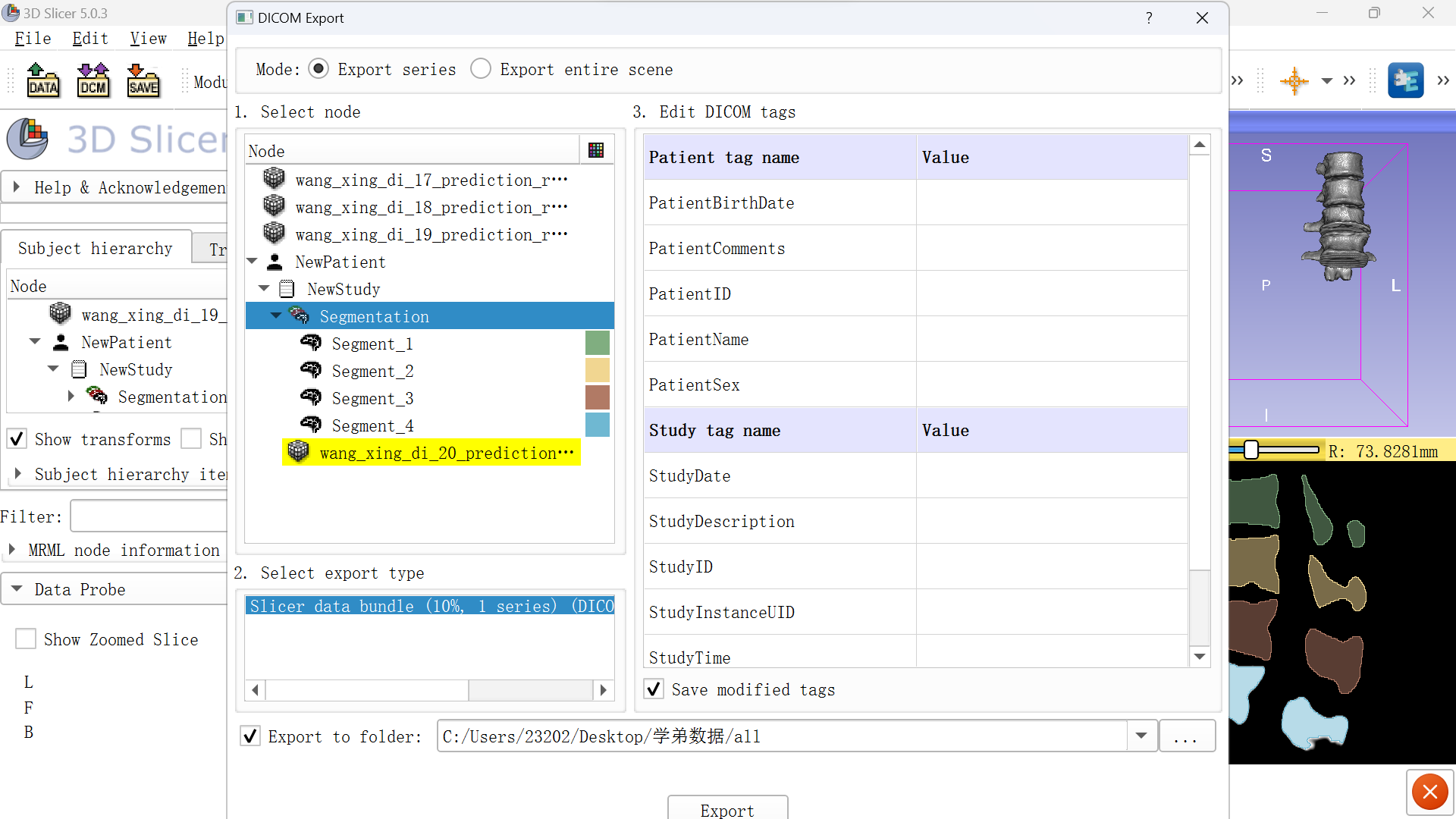
以上完成了多个.nii.gz导为单个.dicom的过程。
2.2.Mimics将.dicom导为.ply格式
下面需要用Mimics导出为.ply格式
2.2.1.加载.dicom文件
打开mimics,点击file,点击New Project wiza,选择需要导入的的dicom文件,点击next导入。
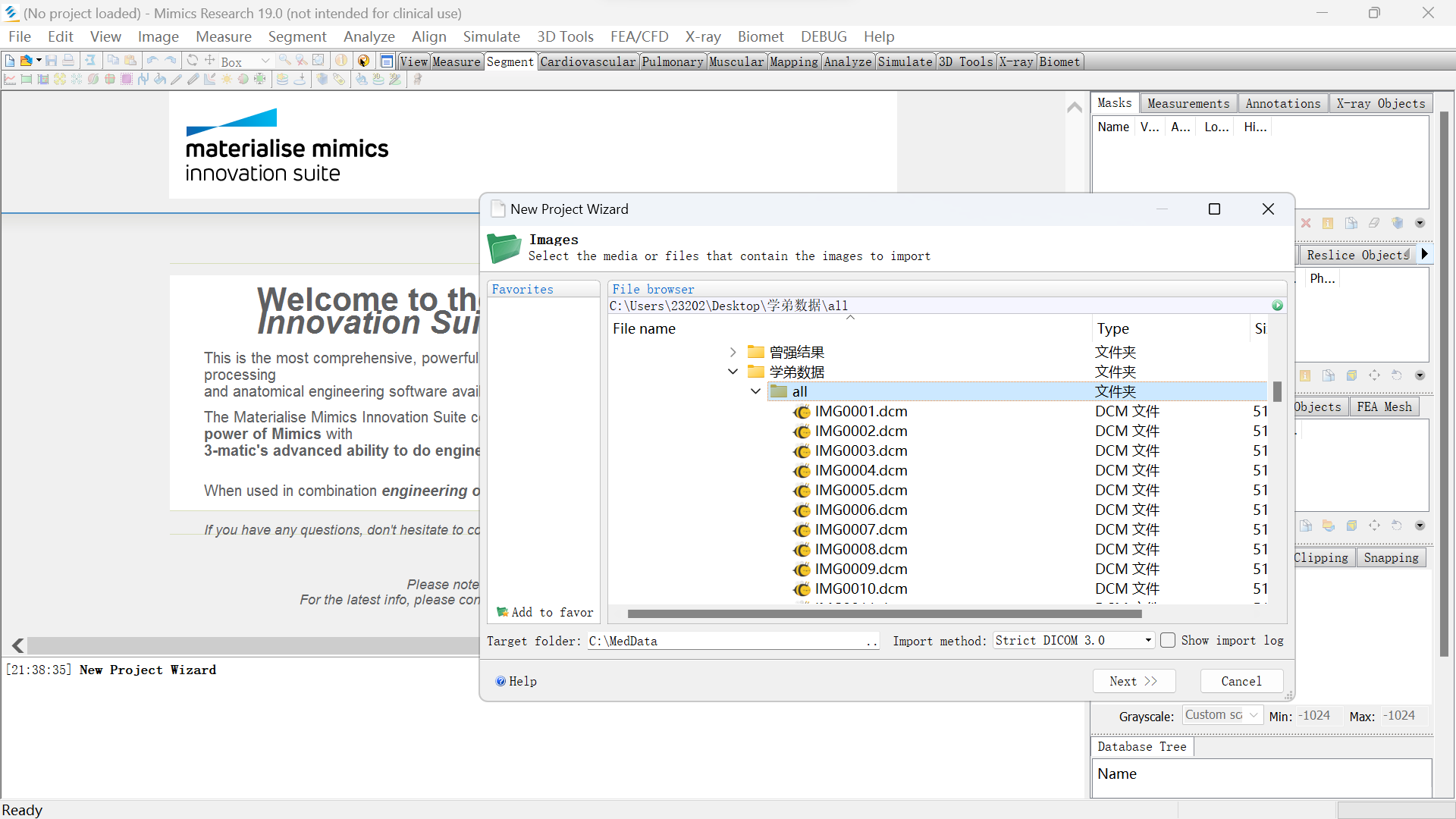
加载进来是这样的。
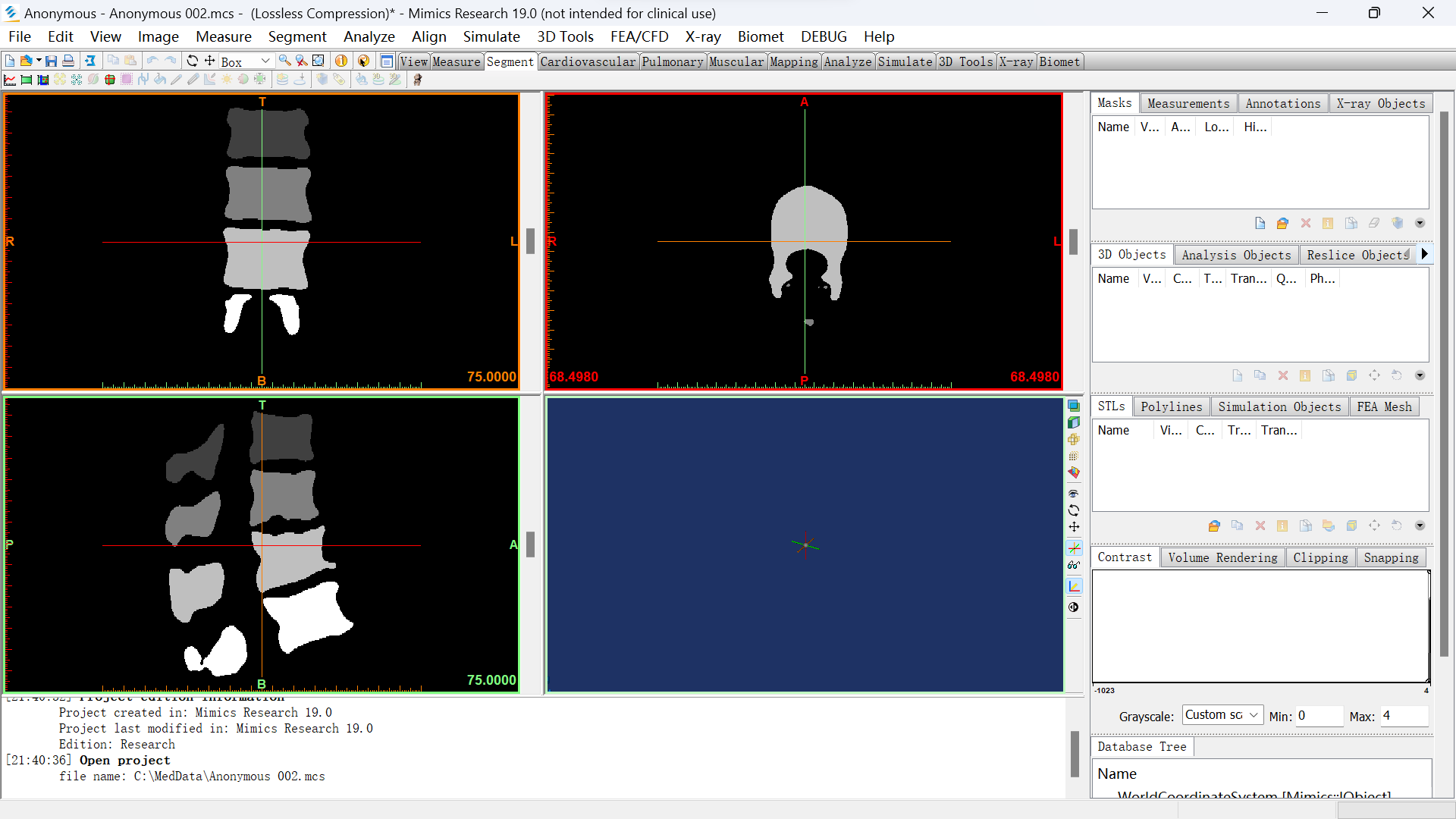
2.2.2.调thresholding
将min设置为1,点击apply。
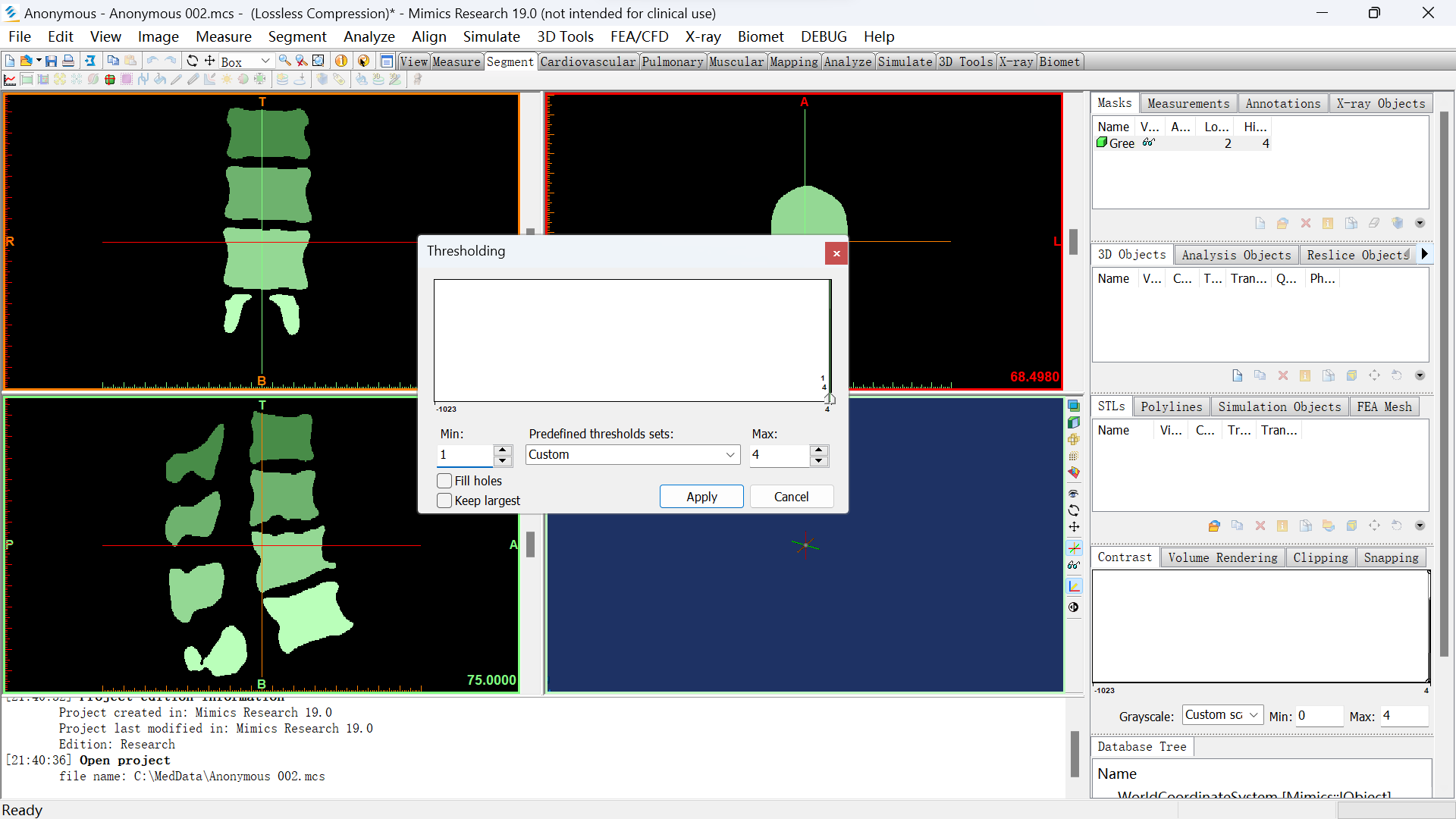
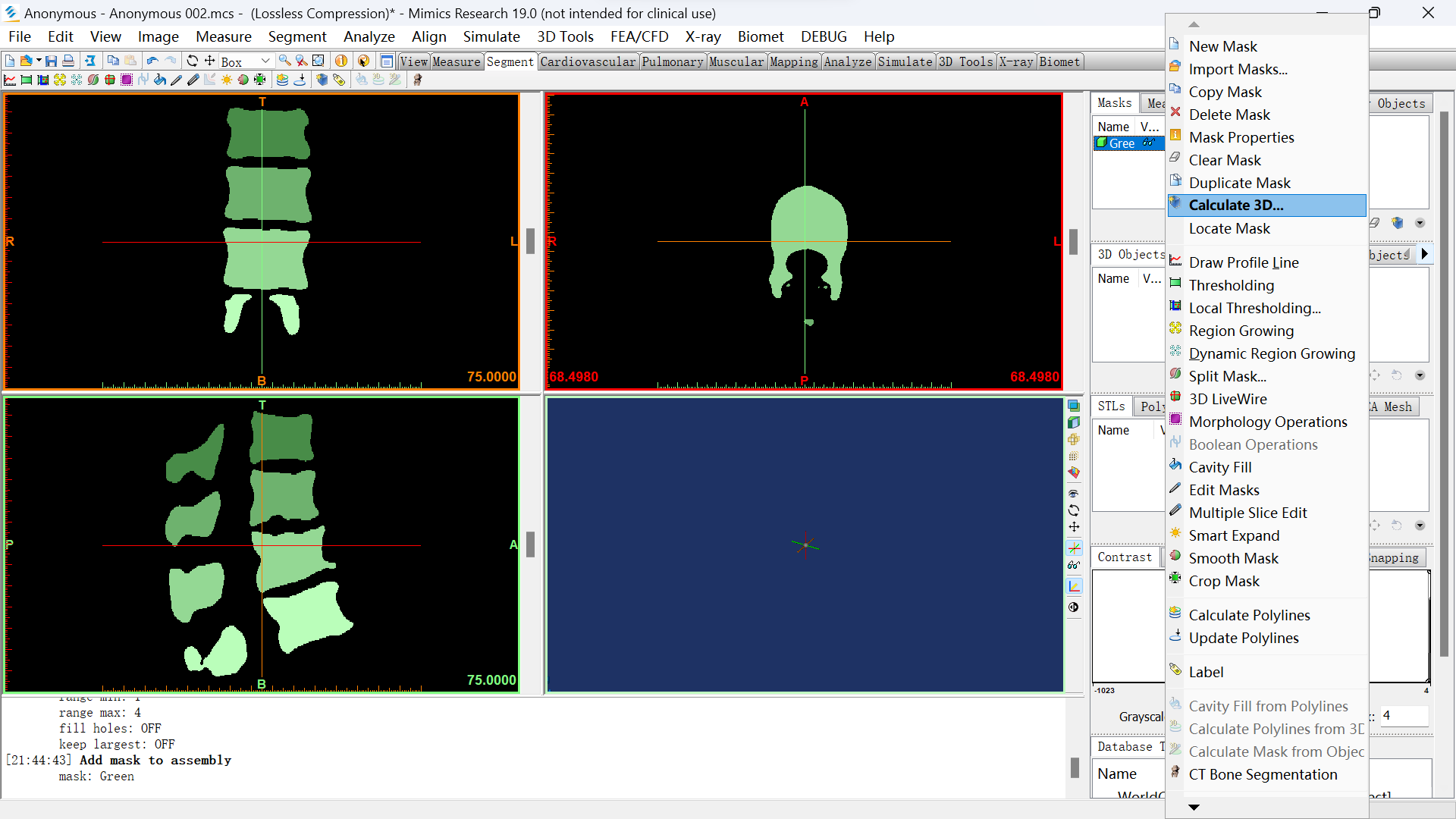
2.2.3.calculate
选择右边的Green右键点击Calulate 3D。
会再右下角的窗口生成三维视图。
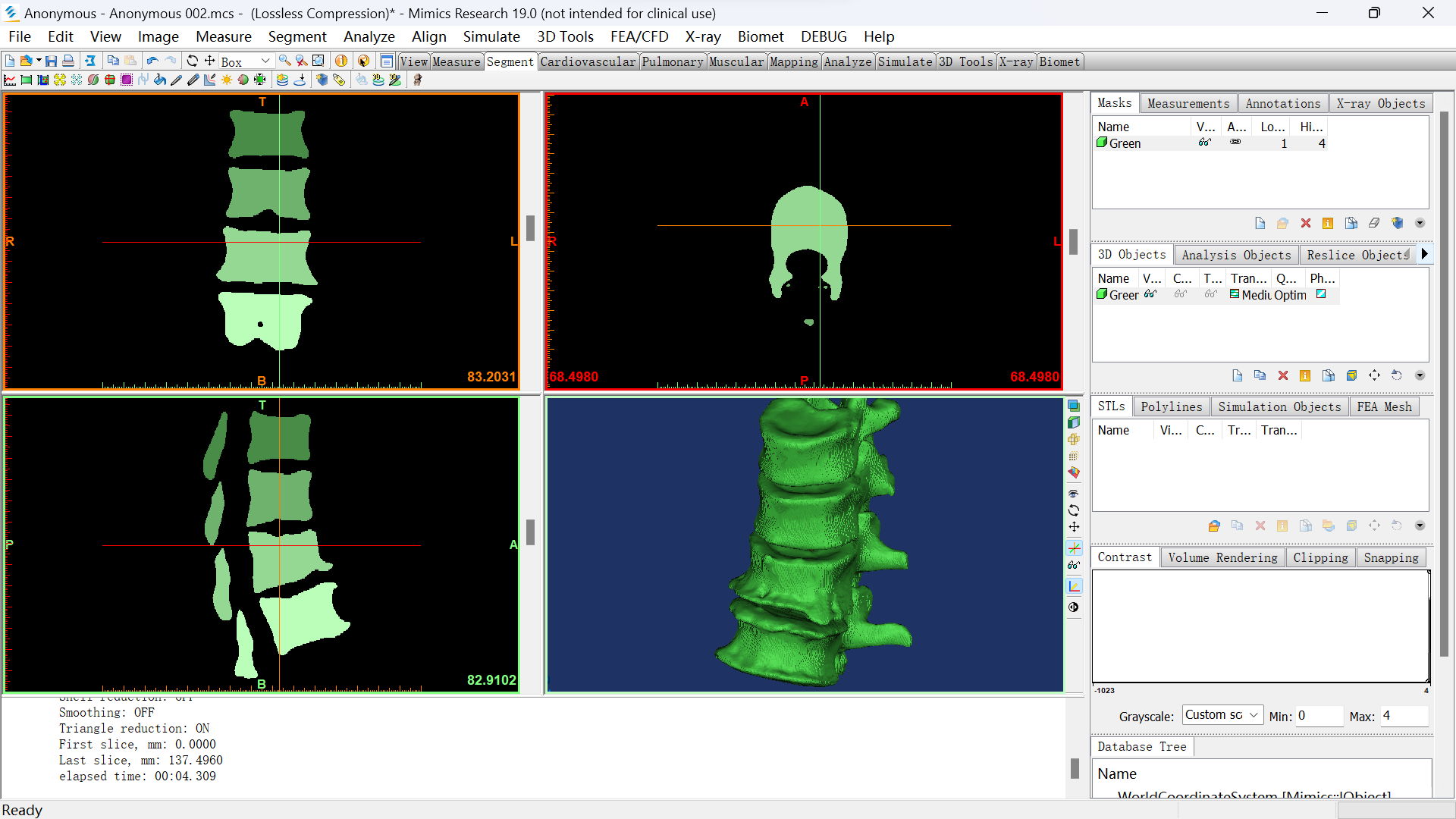
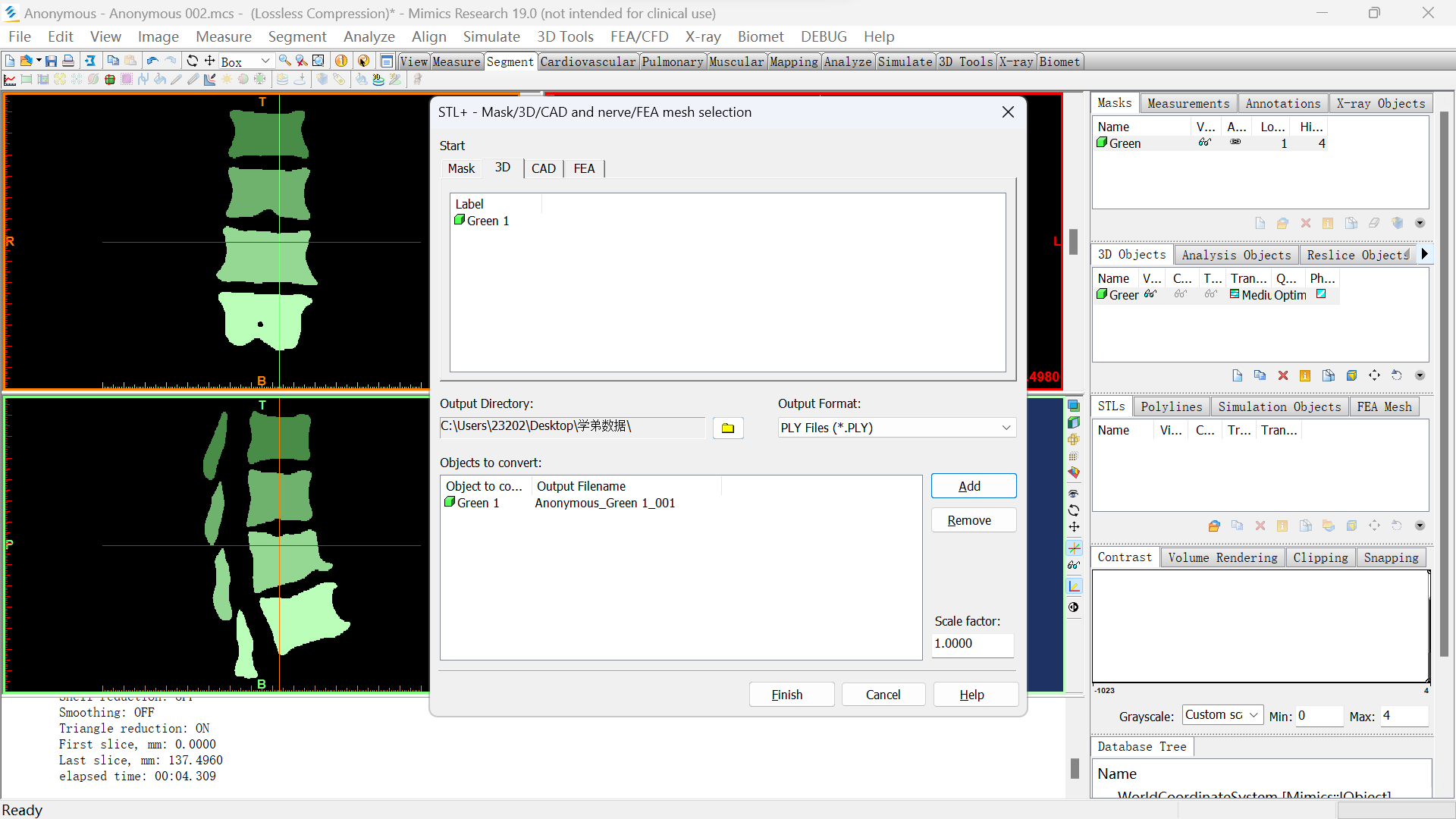
2.2.4.导出ply文件
选择file->export->PLY
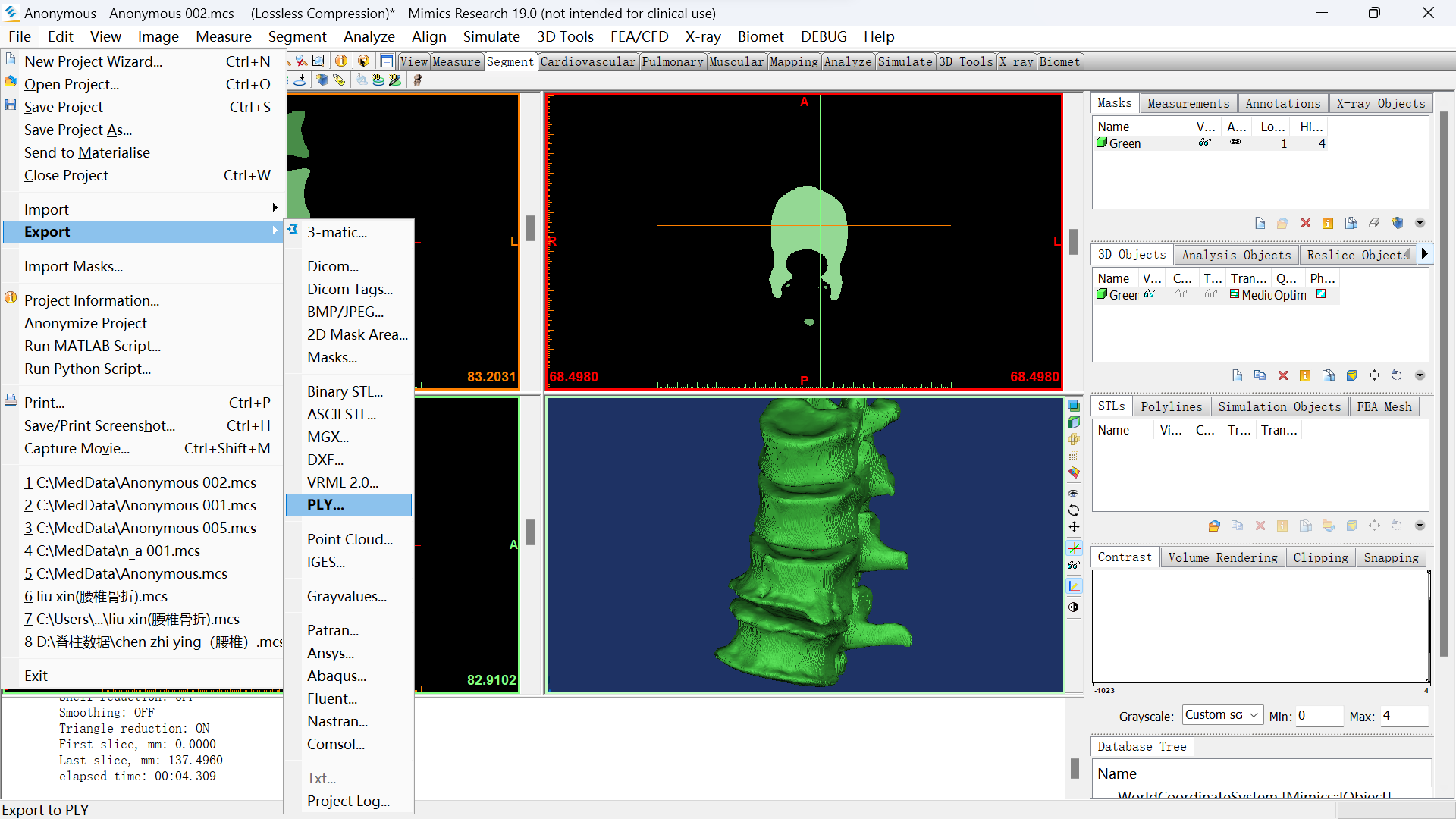
选择3D,点击需要导出的三维文件,再点击add,选择需要导出的文件夹位置即可。
以上即为全部过程,总算把这个过程写下来了,希望可以帮助到需要的朋友。